Giriş / Intoduction
Coronavirüsler (CoV), soğuk algınlığından Orta Doğu Solunum Sendromu (MERS-CoV) ve Şiddetli Akut Solunum Sendromu (SARS-CoV) gibi daha ciddi hastalıklara kadar pek çok hastalığa neden olan büyük bir virüs ailesidir. Bu yeni virüse, yüzey çıkıntıları taç görünümünde olduğu için coronavirus adı verilmiş olup, bu virüs ailesi tek zincirli, pozitif polariteli, zarflı RNA virüsleridir.
Koronavirüs hastalığı (COVID-19), 2019 yılında keşfedilen ve daha önce insanlarda tanımlanmamış yeni bir suştur. Suş, bir bakteri veya virüsün farklı alt türlerinin, aralarında genetik farklılıklar bulunan grupları olarak adlandırılır.
Koronavirüsler zoonotiktir, yani hayvanlar ve insanlar arasında bulaşırlar. Ayrıntılı araştırmalar SARS-CoV’nin misk kedilerinden insanlara ve MERS-CoV’un tek hörgüçlü develerden insanlara bulaştığını göstermiştir. Bilinen birkaç koronavirüs henüz insanları enfekte etmeyen hayvanlarla taşınmaktadır.
Enfeksiyon belirtileri arasında solunum semptomları, ateş, öksürük, nefes darlığı ve solunum güçlüğü bulunur. Daha ciddi vakalarda, enfeksiyon pnömoniye, ciddi akut solunum sendromuna, böbrek yetmezliğine ve hatta ölüme neden olabilir. Solunum sistemi hastalıklarından biri olan pnömoni ya da halk arasında zatürre bakteriler başta olmak üzere mikroorganizmalara bağlı olarak ortaya çıkan akciğer dokusunun iltihaplanmasıdır. Grip gibi bu hastalık da bağışıklık (immun) sistemini baskılayan hastalıklardan biridir. Özellikle ileri yaşlardaki bireylerde hem bağışıklık sisteminin yaşla birlikte yavaşlaması hem de yaşa ve/veya diğer nedenlere bağlı olarak kronik ve diğer hastalıkların görülmesi bu yaşlardaki bireylerde ölüm oranlarını artırabilmektedir. Diğer bir deyişle, bu enfeksiyon komplikasyon yaratarak ve mevcut diğer hastalıkları tetikleyerek bu yaş gruplarında diğer yaş gruplarına göre daha öldürücü olabilir.
Enfeksiyonun yayılmasını önlemek için standart öneriler arasında düzenli el yıkama, öksürme ve hapşırma sırasında ağız ve burnun kapatılması, et ve yumurtaların iyice pişirilmesi yer alır. Öksürme ve hapşırma gibi solunum yolu rahatsızlığı belirtileri gösteren kişilerle yakın temastan kaçınmak bu hastalıktan korunmak için atılacak önemli koruyucu tedbirlerden biridir.
Koronavirüs salgını Çin ile birlikte diğer ülkelere de yayıldığı için Dünya Sağlık Örgütü tarafından 30/01/2020 tarihinde uluslararası halk sağlığı açısından acil durum ilan edilmiştir. Bu hastalıktan bahsedilirken lise ve üniversite yıllarımda benim de ders olarak aldığım Epidemiyoloji biliminden bahsetmemek olmaz. Epidemiyoloji, toplumda sağlıkla ilgili durumların veya olayların (hastalık dahil) dağılımını, görülme sıklıklarını ve bunları etkileyen faktörleri inceleyen bir tıp bilimi dalıdır.
Epidemiyolojik araştırmalar yapmak için çeşitli yöntemler kullanılabilir. Bu yöntemler dağılımı incelemek için sürveyans, tanımlayıcı çalışmalar ve hastalık belirleyicilerini incelemek için analitik çalışmalar olabilir. Bilindiği üzere Koronavirüs hastalığı artık pandemik bir vaka olarak adlandırılmaktadır. Epidemiyolojinin konu alanına giren pandemi dünyada birden fazla ülkede veya kıtada, çok geniş bir alanda
yayılan ve etkisini gösteren salgın hastalıklara verilen genel isim olarak ifade edilmektedir. Diğer bir deyişle, salgın (epidemi) durumundaki bir hastalığın ülke sınırlarını aşarak diğer ülkelere yayılım göstermesidir.
Çalışma kapsamında koronavirüs pandemi vakalarının epidemiyolojik hızlarla birlikte keşifsel veri analizi (EDA) yapılarak vakaların izlediği seyrin ortaya konulması amaçlanmıştır
Metodoloji / Methodology
Çalışmanın amacı, koronavirüs vakalarının epidemiyolojik düzlemde Türkiye’de ve diğer ülkelerde izlediği seyri detaylı bir şekilde keşifsel veri analiziyle ortaya koyarak karar vericilere katkı sunmaktır.
Koronavirüs vakalarının analizinde Türkiye veri setinin oluşturulmasında Türkiye İstatistik Kurumu (TÜİK)’ndan ve Dünya Sağlık Örgütü (DSÖ) raporlarından ve Sağlık Bakanlığı basın açıklamalarından yararlanılmıştır. Bunun nedeni Türkiye veri setine daha derinlemesine bakılmak istenmesidir.
Diğer ülkelere ilişkin koronavirüs vakaları ise Avrupa Birliği kurumlarından biri olan “Avrupa Hastalık Önleme ve Kontrol Merkezi (European Centre for Disease Prevention and Control)” ne ait “https://www.ecdc.europa.eu” web sitesinden alınmıştır.
Analizlerde Türkiye verisi için Microsoft Excel 2016 ve diğer ülke verileri için R programlama dili kullanılmıştır. Şeffaflık olması ve öğretici olması adına bugüne kadar olduğu gibi bu çalışmada da analize ilişkin R kod blokları bu çalışmanın sonunda paylaşılmıştır.
Keşifsel veri analizine geçilmeden önce vaka sayılarından yararlanarak epidemiyolojik hızlar hesaplanmıştır. Bu nedenle, çalışmada kullanılacak hızları açıklamanın faydalı olacağı düşünülmektedir.
Epidemiyoloji biliminde hız kavramı, bir sağlık olayının nüfus içindeki görülme frekansını hesaplamada kullanılan bir ölçüttür. Hızlar, belirli bir zaman diliminde meydana gelen olayların sayısının olayın meydana geldiği nüfusa bölünerek hesaplanır. Eşitliğin payı, sağlık olayı ile karşılaşanların sayısını (X), payda ise risk altındaki nüfusu (X+Y) göstermektedir. Risk altındaki nüfus hastalığa veya sağlık olaylarına karşı duyarlı olan nüfusu ifade etmektedir. Formülize edilirse;
Hız (Rate) = (X / X +Y) * k
Burada, k katsayıyı göstermektedir.
Analizde hızlardan girdi değişkeni olarak kullanılması düşünülen ilk gösterge ölüm veya mortalite ölçütlerinden biri olan Kaba Ölüm Hızı‘dır. Bu hız nüfusun sağlık düzeyini ortaya koymada duyarlı olmasa da bir fikir vermesi açısından önemlidir. Kaba ölüm hızı aşağıdaki eşitlikte gösterilmiştir.
Kaba Ölüm Hızı (Crude Death Rate) = (Bir toplumda belirli bir süre içinde meydana gelen ölüm sayısı / Aynı toplumun aynı süredeki
(yıl ortası) nüfusu) * 1000
Girdi değişkeni olarak kullanılması düşünülen ikinci gösterge ölüm veya fatalite ölçütlerinden biri olan Fatalize Hızı‘dır. Bu gösterge kaba ölüm hızına göre daha duyarlıdır. Diğer bir ifadeyle, hastalığa yakalananların ne kadarının öldüğünü gösterir. Fatalite ise hızı ise şöyledir;
Fatalite Hızı (Fatality Rate) = (Nüfusta belirli bir süre içinde “X” hastalığından ölenlerin sayısı / Aynı nüfusun aynı süre içinde “X” hastalığına yakalananların sayısı) * 100
Morbitide veya hastalık hızları gelişmekte olan hastalığın ortaya çıkma hızını ortaya koymak için kullanılır. Bunlardan biri olan insidans,
bir hastalığın gelişme olasılığını ortaya koymak için kullanılır. Diğer taraftan, bu hızlardan prevelans ise hastalığın görülme sıklığını gösteren bir hızdır. İnsidans ve prevelans da analizde girdi değişkenleri olarak kullanılması düşünülmektedir. İnsidans aşağıdaki eşitlik yardımıyla hesaplanmaktadır.
İnsidans (Incidence) = (Bir nüfusta belirli bir sürede saptanan YENİ vaka sayısı / Aynı toplumun yıl ortası nüfusu) * k
İnsidans hızının kümülatif şekli ise atak hızlarını göstermektedir.
Prevelans ise aşağıdaki eşitlik yardımıyla hesaplanmaktadır.
Prevelans (Prevalence) = (Belirli bir sürede toplam (eski+yeni) vaka sayısı / Risk altındaki kişi sayısı) * k
Keşifsel Veri Analizi Bulguları / Exploratory Data Analysis Findings
Türkiye Örneği / The Case of Turkey
Bu kısımda bahsedilen epidemiyolojik hızların uygulama sonuçlarına yer verilmiştir. İlk olarak günlük onaylanmış vaka sayıları ile ölüm vaka sayıları Tablo 1’de verilmiştir. Tablo 1’e göre öne çıkan bulgular şöyledir:
- Toplam vaka sayısı 3629’a ulaştığı 26.03.2020 tarihinde görülen yeni vaka sayısı 1196 olup, bu sayı Türkiye’de salgının başladığı günden bugüne kadar en yüksek vaka sayısıdır.
- Kümülatif vaka sayısından kümülatif ölen kişi sayısı çıkarılmasından sonra kalan toplam aktif vaka sayısı 3450’dir.
- Salgından ölen toplam kişi sayısı 59 olup, en yüksek ölüm vakalarının yaşandığı gün 26.03.2020 tarihidir.
Tablo 1. Koronavirüs Vaka Sayıları / Number of Coronavirus Cases, Turkey

Epidemiyolojik hızları gösteren bulgular ise Tablo 2’de sunulmuştur. Tablo 2’ye göre öne çıkan bulgular şöyledir:
- Sağlık düzeyini belirlemede duyarlı bir gösterge olmasa da fikir vermesi açısından kaba ölüm hızı hesaplanmıştır. Buna göre kaba ölüm hızı en yüksek seviyesine ulaştığı 25.3.2020 tarihinde milyonda 0,19’dur. Katsayı 1000 yerine milyon alınmasının nedeni ortaya konulan bulgunun çok küçük değer almasından kaynaklanmaktadır. Toplam ölen kişi sayına göre kaba ölüm hızı ise milyonda yaklaşık 1’tir.
- 60 ve üzeri yaş üzerindekiler yüksek risk grubuna girdiği için ve genellikle açıklanan ölüm sayıları bu yaş gruba ait olduğu için varsayımsal olarak bu yaş gruba özel kaba ölüm hızı da hesaplanmıştır. Bu yaş grubunda hesaplanan kaba ölüm hızı milyonda yaklaşık 1,4’tür. Toplam ölen kişi sayına göre bu yaş grubuna özel kaba ölüm hızı ise milyonda 6,8’dir.
- Fatalite hızının en yüksek olduğu tarih binde yaklaşık 43 ile 21.3.2020 tarihidir. Toplam vaka sayılarına göre fatalite hızı ise binde yaklaşık 21’dir.
- Yapılan test sayısı 40290, onaylanmış vaka sayısı 3629’dur. Belirli bir zaman diliminde bir hastalığın bir nüfus grubunda görülme sıklığını ortaya koyan prevelans hızı % 9’dur. Buradan salgın meydana geldiği süre içerisinde her 100 vakadan 9’unda koronavirüs vakası tespit edildiği anlamı çıkmaktadır.
Tablo 2. Koronavirüs Vakalarının Epidemiyolojik Hızları / Epidemiological Rates of Coronavirus Cases, Turkey

Dünya ve Diğer Ülke Örnekleri / Cases of World and Other Countries
İlk olarak adım adım uygulamaya geçilerek Avrupa Hastalık Önleme ve Kontrol Merkezi‘nin web sitesinden xlsx (Microsoft Excel tarafından kullanılan dosya formatı) uzantılı olarak okutulan onaylanmış vaka ve ölüm sayıları veri seti tanıtılmıştır.
Çalışmanın bu kısmında epidemiyolojik hızlarla birlikte keşifsel veri analizi yapılan ülkeler İtalya, Fransa, Almanya, Güney Kore, İspanya ve ABD, İngiltere’dir.
Aşağıda yazdığım R kod bloğu ile birlikte veri seti Avrupa Hastalık Önleme ve Kontrol Merkezi veri tabanından indirilmeden güncel veri akışı sağlanabilecektir.
url = "https://www.ecdc.europa.eu/sites/default/files/documents/COVID-19-geographic-disbtribution-worldwide-2020-03-25.xlsx"
destfile <- "COVID_19_geographic_disbtribution_worldwide_2020_03_25.xlsx"
curl::curl_download(url, destfile)
ECDC_Data <- read_excel(destfile)
Data<- ECDC_Data[,-8:-10]
df<-Data %>% rename(Country="Countries and territories", Date="DateRep")
df
#Eksik veri olup olmadığının gözden geçirilmesi için
sum(is.na(df$Cases))#0
Veri setinde ihtiyaç duyulmayan son 3 sütun çıkarılmıştır. Bu işlemden sonra veri setinde kalan değişken sayısı 7’dir. Veri setindeki toplam gözlem sayısı ise 6931’dir. Aşağıda mevcut değişkenler, değişkenin veri tipi ile birlikte gözlem sayısı görülmektedir.
Classes ‘tbl_df’, ‘tbl’ and 'data.frame': 6931 obs. of 7 variables:
$ Date : POSIXct, format: "2020-03-26" "2020-03-25" ...
$ Day : num 26 25 24 23 22 21 20 19 18 17 ...
$ Month : num 3 3 3 3 3 3 3 3 3 3 ...
$ Year : num 2020 2020 2020 2020 2020 2020 2020 2020 2020 2020 ...
$ Cases : num 33 2 6 10 0 2 0 0 1 5 ...
$ Deaths : num 0 0 1 0 0 0 0 0 0 0 ...
$ Country: chr "Afghanistan" "Afghanistan" "Afghanistan" "Afghanistan" ...
Veri setinde koronavirüs salgınının yaşandığı aşağıdaki listesi verilen 192 ülke ve/veya bölge bulunmaktadır.
[1] "Afghanistan"
[2] "Angola"
[3] "Albania"
[4] "Andorra"
[5] "Argentina"
[6] "Antigua_and_Barbuda"
[7] "Algeria"
[8] "Armenia"
[9] "Aruba"
[10] "Australia"
[11] "Austria"
[12] "Azerbaijan"
[13] "Bahamas"
[14] "Bahrain"
[15] "Bangladesh"
[16] "Barbados"
[17] "Belarus"
[18] "Belgium"
[19] "Belize"
[20] "Benin"
[21] "Bermuda"
[22] "Bhutan"
[23] "Bolivia"
[24] "Bosnia_and_Herzegovina"
[25] "Brazil"
[26] "Brunei_Darussalam"
[27] "Bulgaria"
[28] "Burkina_Faso"
[29] "Cambodia"
[30] "Cameroon"
[31] "Canada"
[32] "Cape_Verde"
[33] "Cases_on_an_international_conveyance_Japan"
[34] "Cayman_Islands"
[35] "Central_African_Republic"
[36] "Chad"
[37] "Chile"
[38] "China"
[39] "Colombia"
[40] "Congo"
[41] "Costa_Rica"
[42] "Cote_dIvoire"
[43] "Croatia"
[44] "Cuba"
[45] "Curaçao"
[46] "Cyprus"
[47] "Czech_Republic"
[48] "Democratic_Republic_of_the_Congo"
[49] "Denmark"
[50] "Djibouti"
[51] "Dominica"
[52] "Dominican_Republic"
[53] "Ecuador"
[54] "Egypt"
[55] "El_Salvador"
[56] "Equatorial_Guinea"
[57] "Eritrea"
[58] "Estonia"
[59] "Eswatini"
[60] "Ethiopia"
[61] "Faroe_Islands"
[62] "Fiji"
[63] "Finland"
[64] "France"
[65] "French_Polynesia"
[66] "Gabon"
[67] "Gambia"
[68] "Georgia"
[69] "Germany"
[70] "Ghana"
[71] "Gibraltar"
[72] "Greece"
[73] "Greenland"
[74] "Grenada"
[75] "Guam"
[76] "Guatemala"
[77] "Guernsey"
[78] "Guinea"
[79] "Guyana"
[80] "Haiti"
[81] "Holy_See"
[82] "Honduras"
[83] "Hungary"
[84] "Iceland"
[85] "India"
[86] "Indonesia"
[87] "Iran"
[88] "Iraq"
[89] "Ireland"
[90] "Isle_of_Man"
[91] "Israel"
[92] "Italy"
[93] "Jamaica"
[94] "Japan"
[95] "Jersey"
[96] "Jordan"
[97] "Kazakhstan"
[98] "Kenya"
[99] "Kosovo"
[100] "Kuwait"
[101] "Kyrgyzstan"
[102] "Laos"
[103] "Latvia"
[104] "Lebanon"
[105] "Liberia"
[106] "Libya"
[107] "Liechtenstein"
[108] "Lithuania"
[109] "Luxembourg"
[110] "Madagascar"
[111] "Malaysia"
[112] "Maldives"
[113] "Mali"
[114] "Malta"
[115] "Mauritania"
[116] "Mauritius"
[117] "Mexico"
[118] "Moldova"
[119] "Monaco"
[120] "Mongolia"
[121] "Montenegro"
[122] "Montserrat"
[123] "Morocco"
[124] "Mozambique"
[125] "Myanmar"
[126] "Namibia"
[127] "Nepal"
[128] "Netherlands"
[129] "New_Caledonia"
[130] "New_Zealand"
[131] "Nicaragua"
[132] "Niger"
[133] "Nigeria"
[134] "North_Macedonia"
[135] "Norway"
[136] "Oman"
[137] "Pakistan"
[138] "Palestine"
[139] "Panama"
[140] "Papua_New_Guinea"
[141] "Paraguay"
[142] "Peru"
[143] "Philippines"
[144] "Poland"
[145] "Portugal"
[146] "Qatar"
[147] "Romania"
[148] "Russia"
[149] "Rwanda"
[150] "Saint_Kitts_and_Nevis"
[151] "Saint_Lucia"
[152] "Saint_Vincent_and_the_Grenadines"
[153] "San_Marino"
[154] "Saudi_Arabia"
[155] "Senegal"
[156] "Serbia"
[157] "Seychelles"
[158] "Singapore"
[159] "Sint_Maarten"
[160] "Slovakia"
[161] "Slovenia"
[162] "Somalia"
[163] "South_Africa"
[164] "South_Korea"
[165] "Spain"
[166] "Sri_Lanka"
[167] "Sudan"
[168] "Suriname"
[169] "Sweden"
[170] "Switzerland"
[171] "Syria"
[172] "Taiwan"
[173] "Thailand"
[174] "Timor_Leste"
[175] "Togo"
[176] "Trinidad_and_Tobago"
[177] "Tunisia"
[178] "Turkey"
[179] "Turks_and_Caicos_islands"
[180] "Uganda"
[181] "Ukraine"
[182] "United_Arab_Emirates"
[183] "United_Kingdom"
[184] "United_Republic_of_Tanzania"
[185] "United_States_of_America"
[186] "United_States_Virgin_Islands"
[187] "Uruguay"
[188] "Uzbekistan"
[189] "Venezuela"
[190] "Vietnam"
[191] "Zambia"
[192] "Zimbabwe"
Mevcut veri setinde aktif vaka sayıları ile birlikte fatalite hızları da hesaplandığı için değişken sayısı 7’den 9’a çıkarılmıştır.
Dünya / World
Dünyadaki Vaka Sayıları İle Birlikte Fatalite Hızları /Fatality Rates with Case Numbers in the World
Ortaya konulan fatalite hızı, Dünyada koronavirüs hastalığına yakalanan her 1000 kişinden yaklaşık 45’inin hayatını kaybettiğini göstermektedir.

Dünyadaki Aylık Fatalite Hızları / Monthly Fatality Rates in the World

26.3.2020 Tarihinde Fatalite Hızının En Yüksek Olduğu İlk 12 Ülke / Top 12 Countries with the Highest Fatality Rate on March 26, 2020

Dünyada Kümülatif Fatalite Hızının En Yüksek Olduğu 12 Ülke/Bölge // Top 12 Countries with the Highest Cumulative Fatality Rate in the World on March 26, 2020
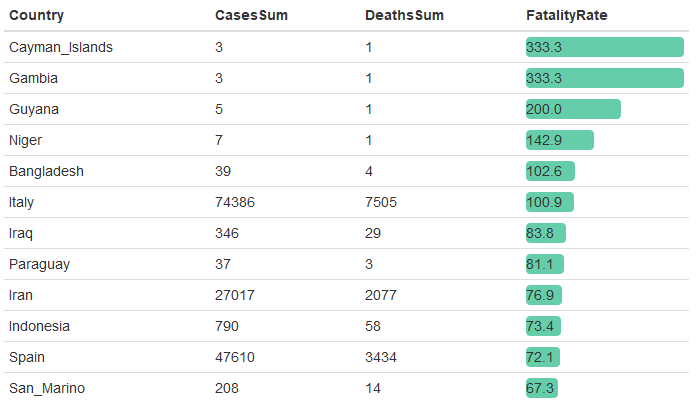
Dünyada Kümülatif Fatalite Hızının En Düşük Olduğu 12 Ülke/Bölge / Top 12 Countries / Regions with the Lowest Cumulative Fatality Rate in the World

Dünyadaki Vaka Sayılarının Aylara Göre Dağılımı /Distribution of Case Numbers in the World by Month

Dünyadaki Ölüm Vakalarının Aylara Göre Dağılımı / Distribution of Death Cases in the World by Month

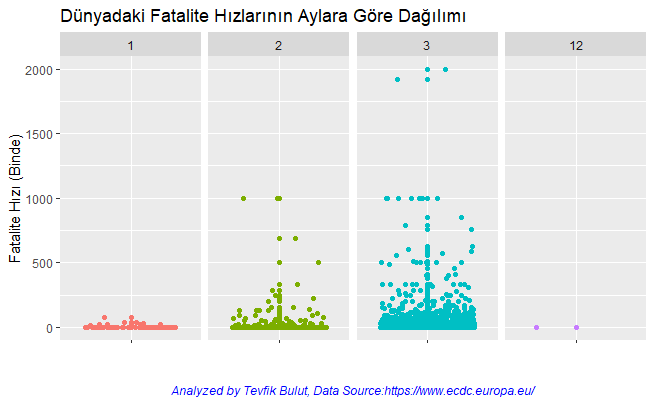
Dünyadaki Fatalite Hızlarının Aylara Göre Dağılımı / Distribution of Fatality Rates in the World by Month
İtalya / Italy
İtalya’daki Fatalite Hızlarının Aylara Göre Dağılımı / Distribution of Fatality Rates in Italy by Month

İtalya’daki Aylık Fatalite Hızları / Monthly Fatality Rates in Italy
Ortaya konulan fatalite hızlarına göre 3. ayda İtalya’da Koronavirüs hastalığına yakalanan her 1000 kişinden yaklaşık 102’sinin hayatını kaybettiğini ortaya koymaktadır.

İspanya / Spain
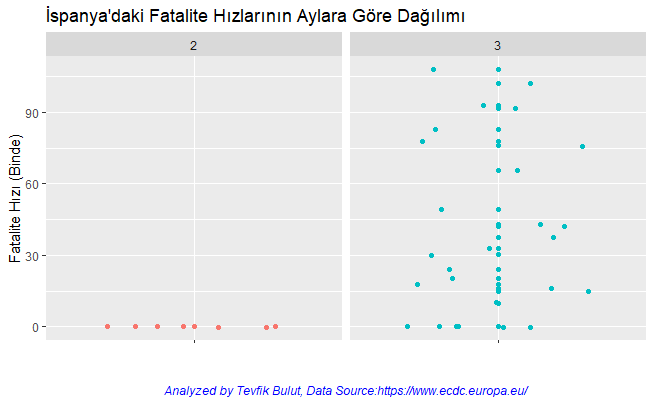
İspanya’daki Fatalite Hızlarının Aylara Göre Dağılımı / Distribution of Fatality Rates in Spain by Month
İspanya’daki Aylık Fatalite Hızları / Monthly Fatality Rates in Spain
Ortaya konulan fatalite hızlarına göre 3. ayda İspanya’da Koronavirüs vakaları çok ani ve keskin bir yükseliş göstermiştir. Fatalize hızları, Koronavirüs hastalığına yakalanan her 1000 kişinden 72’sinin hayatını kaybettiğini ortaya koymaktadır.

Fransa / France
Fransa’daki Fatalite Hızlarının Aylara Göre Dağılımı / Distribution of Fatality Rates in France by Month

Fransa’daki Aylık Fatalite Hızları / Monthly Fatality Rates in France
Ortaya konulan fatalite hızlarına göre 3. ayda Fransa’da Koronavirüs hastalığına yakalanan her 1000 kişinden yaklaşık 53’sinin hayatını kaybettiğini ortaya koymaktadır.

Güney Kore / South Korea
Güney Kore’deki Fatalite Hızlarının Aylara Göre Dağılımı / Distribution of Fatality Rates in South Korea by Month

Güney Korede’ki Aylık Fatalite Hızları / Monthly Fatality Rates in South Korea
Ortaya konulan fatalite hızlarına göre 3. ayda Güney Kore’de Koronavirüs hastalığına yakalanan her 1000 kişinden yaklaşık 18’inin hayatını kaybettiğini ortaya koymaktadır.

Almanya / Germany
Almanya’daki Fatalite Hızlarının Aylara Göre Dağılımı / Distribution of Fatality Rates in Germany by Month

Almanya’daki Aylık Fatalite Hızları / Monthly Fatality Rates in Germany
Ortaya konulan fatalite hızlarına göre 3. ayda Almanya’da Koronavirüs hastalığına yakalanan her 1000 kişinden 5’inin hayatını kaybettiğini ortaya koymaktadır.
ABD / USA
Amerika’daki Fatalite Hızlarının Aylara Göre Dağılımı / Distribution of Fatality Rates in USA by Month

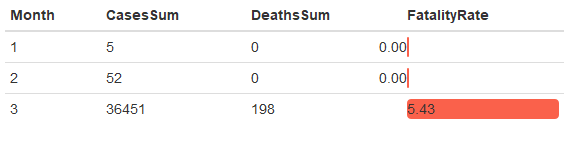
ABD’deki Aylık Fatalite Hızları / Monthly Fatality Rates in USA
Ortaya konulan fatalite hızlarına göre 3. ayda ABD’de Koronavirüs hastalığına yakalanan her 1000 kişinden 15’inin hayatını kaybettiğini ortaya koymaktadır.

İngiltere / UK
İngiltere’deki Fatalite Hızlarının Aylara Göre Dağılımı / Distribution of Fatality Rates in UK by Month
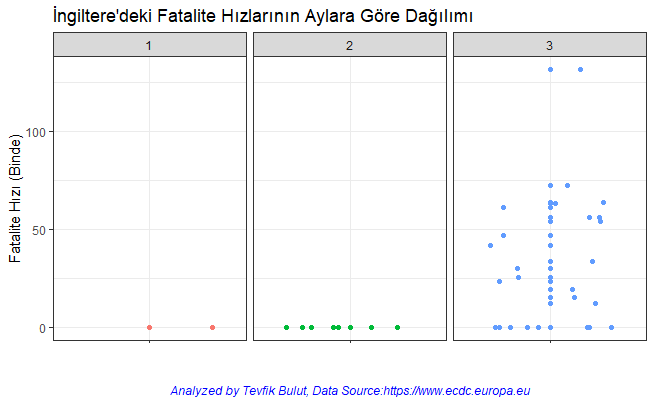
İngiltere’deki Aylık Fatalite Hızları / Monthly Fatality Rates in UK
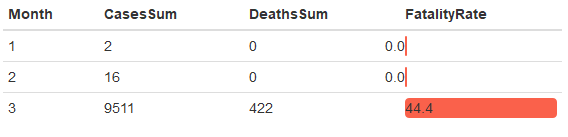
Ortaya konulan fatalite hızlarına göre 3. ayda İngiltere’de Koronavirüs hastalığına yakalanan her 1000 kişinden 15’inin hayatını kaybettiğini ortaya koymaktadır.
Sonuç / Conclusion
Özetle, Avrupa Hastalık Önleme ve Kontrol Merkezi‘nden alınan koronavirüs hastalık veri setinin epidemiyolojik hızlarla birlikte keşifsel veri analizi yapılarak koronavirüs pandemisinin trendi hakkında farkındalık oluşturulması amaçlanmıştır.
Faydalı olması ve farkındalık oluşturması dileğiyle.
Bilimle ve teknolojiyle kalınız.
Saygılarımla.
Note: It can not be cited or copied without referencing.
Not: Kaynak gösterilmeden alıntı yapılamaz veya kopyalanamaz.
Yararlanılan Kaynaklar / Resources Utilized
- World Health Organization (2020). Report of the WHO-China Joint Mission on Coronavirus Disease 2019 (COVID-19). Available online at: https://www.who.int/docs/default-source/coronaviruse/who-china-joint-mission-on-covid-19-final-report.pdf
- https://www.who.int/health-topics/coronavirus
- https://www.ecdc.europa.eu/en/publications-data/download-todays-data-geographic-distribution-covid-19-cases-worldwide
- http://www.hastane.hacettepe.edu.tr/_948.html
- https://www.youtube.com/watch?v=mOV1aBVYKGA&t=42s
- Humanitarian Data Exchange (HDX), https://data.humdata.org/. 19.03.2020
- http://www.halksagligi.hacettepe.edu.tr/sunumlar_ve_seminerler/OLCUTLERTB18MART2015.pdf
- https://www.youtube.com/watch?v=4KFP3tay5vM
- De Livera, A. M., Hyndman, R. J., & Snyder, R. D. (2011). Forecasting time series with complex seasonal patterns using exponential smoothing. J American Statistical Association, 106(496), 1513–1527. https://robjhyndman.com/publications/complex-seasonality/
- http://tagteam.harvard.edu/hub_feeds/1981/feed_items/273923
- https://robjhyndman.com/papers/ComplexSeasonality.pdf
- https://www.medicalpark.com.tr/zaturre-nedir-belirtileri-ve-tedavileri-nelerdir/hg-152
- https://www.who.int/topics/epidemiology/en/
- https://www.grip.gov.tr/depo/saglik-calisanlari/ulusal_pandemi_plani.pdf
- https://www.r-project.org/
- https://www.who.int/docs/default-source/coronaviruse/getting-workplace-ready-for-covid-19.pdf
Analizde Kullanılan R Programlama Dili Kodları / R Programming Language Codes Used in Analysis
Yapılan analizlerde kullanılan R programlama diline ait kodlar aşağıda yer almaktadır.
sapply(c("dplyr","tibble","tidyr","ggplot2","formattable","ggthemes","xts","zoo","stringr","lubridate","readxl","xlsx"), require, character.only = TRUE)
url = "https://www.ecdc.europa.eu/sites/default/files/documents/COVID-19-geographic-disbtribution-worldwide-2020-03-25.xlsx"
destfile <- "COVID_19_geographic_disbtribution_worldwide_2020_03_25.xlsx"
curl::curl_download(url, destfile)
ECDC_Data <- read_excel(destfile)
Data<- ECDC_Data[,-8:-10]
df<-Data %>% rename(Country="Countries and territories", Date="DateRep")
df
#Eksik veri olup olmadığının gözden geçirilmesi için
sum(is.na(df$Cases))#0
str(df)
unique(df$Country)
world<-df %>%
filter(Cases>0) %>%
group_by(Date, Month, Country, Cases, Deaths) %>%
mutate(ActiveCases=Cases-Deaths, FatalityRate=(Deaths/Cases)*1000)
%>%
arrange(desc(Date))
world<- as_tibble(world)
worldfr<-select(world, Cases, Deaths) %>% summarise(CasesSum = sum(Cases), DeathsSum = sum(Deaths), FatalityRate= sum(Deaths)/sum(Cases)*1000)
formattable(worldfr)
worldfrmonth<-select(world, Month, Cases, Deaths) %>%
group_by(Month) %>%
summarise(CasesSum = sum(Cases), DeathsSum = sum(Deaths), FatalityRate= round(sum(Deaths)/sum(Cases)*1000,1))
formattable(worldfrmonth,
align = rep("l",4),
list('Month' = formatter("span", style = ~ style(color = "grey", font.weight = "bold")),
'FatalityRate' = color_bar("#FA614B")))
topday<-df %>%
filter(Cases>0, Date==as.Date("2020-03-26")) %>%
group_by(Date, Month, Country, Cases, Deaths) %>%
mutate(ActiveCases=Cases-Deaths, FatalityRate=(Deaths/Cases)*1000) %>%
arrange(desc(Month))
topday<- as_tibble(topday)
topday
topdayfr<-select(topday, Country, Cases, Deaths) %>%
group_by(Country) %>%
summarise(CasesSum = sum(Cases), DeathsSum = sum(Deaths), FatalityRate= round((sum(Deaths)/sum(Cases)*1000),1)) %>%
arrange(desc(FatalityRate)) %>%
top_n(12)
topdayfr
formattable(head(topdayfr,12),
align = rep("l",4),
list('Date' = formatter("span", style = ~ style(color = "grey", font.weight = "bold")),
'FatalityRate' = color_bar("#00BFFF")))
world %>% ggplot(aes("", Cases, col=as.factor(Month)))+
geom_point()+
geom_jitter()+
facet_grid(~Month, scales="free")+
labs(title="Dünyadaki Vakaların Aylar İçindeki Dağılımı")+
xlab("") + ylab("Vaka Sayısı")+
labs(caption = "Analyzed by Tevfik Bulut, Data Source:https://www.ecdc.europa.eu/, 2020-03-25")+
theme(plot.caption = element_text(color = "blue", face="italic", hjust=0.5))+
theme(legend.position = "none")
world %>% ggplot(aes("", Deaths, color=as.factor(Month)))+
geom_point()+
geom_jitter()+
facet_grid(~Month, scales="free")+
labs(title="Dünyadaki Ölüm Vakalarının Aylar İçindeki Dağılımı")+
xlab("") + ylab("Ölüm Sayısı")+
labs(caption = "Analyzed by Tevfik Bulut, Data Source:https://www.ecdc.europa.eu/, 2020-03-25")+
theme(plot.caption = element_text(color = "blue", face="italic", hjust=0.5)) +
theme(legend.position = "none")
world %>% ggplot(aes("", FatalityRate, color=as.factor(Month)))+
geom_point()+
geom_jitter()+
facet_grid(~Month, scales="free")+
labs(title="Dünyadaki Fatalite Hızlarının Aylara Göre Dağılımı")+
xlab("") + ylab("Fatalite Hızı (Binde)")+
labs(caption = "Analyzed by Tevfik Bulut, Data Source:https://www.ecdc.europa.eu/")+
theme(plot.caption = element_text(color = "blue", face="italic", hjust=0.5)) +
theme(legend.position = "none")
italy<-df %>%
filter(Cases>0, Country=="Italy") %>%
group_by(Date, Month, Country, Cases, Deaths) %>%
mutate(ActiveCases=Cases-Deaths, FatalityRate=(Deaths/Cases)*1000) %>%
arrange(desc(Date))
italy<- as_tibble(italy)
italy
italy %>% ggplot(aes("", FatalityRate, color=as.factor(Month)))+
geom_point()+
geom_jitter()+
facet_grid(~Month, scales="free")+
labs(title="İtalya'daki Fatalite Hızlarının Aylara Göre Dağılımı")+
xlab("") + ylab("Fatalite Hızı (Binde)")+
labs(caption = "Analyzed by Tevfik Bulut, Data Source:https://www.ecdc.europa.eu/")+
theme(plot.caption = element_text(color = "blue", face="italic", hjust=0.5)) +
theme(legend.position = "none")
italyfrmonth<-select(italy, Month, Cases, Deaths) %>%
group_by(Month) %>%
summarise(CasesSum = sum(Cases), DeathsSum = sum(Deaths), FatalityRate= sum(Deaths)/sum(Cases)*1000)
formattable(italyfrmonth,
align = rep("l",4),
list('Date' = formatter("span", style = ~ style(color = "grey", font.weight = "bold")),
'FatalityRate' = color_bar("#FA614B")))
spain<-df %>%
filter(Cases>0, Country=="Spain") %>%
group_by(Date, Month, Country, Cases, Deaths) %>%
mutate(ActiveCases=Cases-Deaths, FatalityRate=(Deaths/Cases)*1000) %>%
arrange(desc(Date))
spain<- as_tibble(spain)
spain
spain %>% ggplot(aes("", FatalityRate, color=as.factor(Month)))+
geom_point()+
geom_jitter()+
facet_grid(~Month, scales="free")+
labs(title="İspanyadaki Fatalite Hızlarının Aylara Göre Dağılımı")+
xlab("") + ylab("Fatalite Hızı (Binde)")+
labs(caption = "Analyzed by Tevfik Bulut, Data Source:https://www.ecdc.europa.eu/")+
theme(plot.caption = element_text(color = "blue", face="italic", hjust=0.5)) +
theme(legend.position = "none")
spainfrmonth<-select(spain, Month, Cases, Deaths) %>%
group_by(Month) %>%
summarise(CasesSum = sum(Cases), DeathsSum = sum(Deaths), FatalityRate= sum(Deaths)/sum(Cases)*1000)
formattable(spainfrmonth,
align = rep("l",4),
list('Date' = formatter("span", style = ~ style(color = "grey", font.weight = "bold")),
'FatalityRate' = color_bar("#FA614B")))
france<-df %>%
filter(Cases>0, Country=="France") %>%
group_by(Date, Month, Country, Cases, Deaths) %>%
mutate(ActiveCases=Cases-Deaths, FatalityRate=(Deaths/Cases)*1000) %>%
arrange(desc(Date))
france<- as_tibble(france)
france
france %>% ggplot(aes("", FatalityRate, color=as.factor(Month)))+
geom_point()+
geom_jitter()+
facet_grid(~Month, scales="free")+
labs(title="Fransa'daki Fatalite Hızlarının Aylara Göre Dağılımı")+
xlab("") + ylab("Fatalite Hızı (Binde)")+
labs(caption = "Analyzed by Tevfik Bulut, Data Source:https://www.ecdc.europa.eu/, 2020-03-25")+
theme(plot.caption = element_text(color = "blue", face="italic", hjust=0.5)) +
theme(legend.position = "none")
francefrmonth<-select(france, Month, Cases, Deaths) %>%
group_by(Month) %>%
summarise(CasesSum = sum(Cases), DeathsSum = sum(Deaths), FatalityRate= sum(Deaths)/sum(Cases)*1000)
formattable(francefrmonth,
align = rep("l",4),
list('Date' = formatter("span", style = ~ style(color = "grey", font.weight = "bold")),
'FatalityRate' = color_bar("#FA614B")))
South_Korea<-df %>%
filter(Cases>0, Country=="South_Korea") %>%
group_by(Date, Month, Country, Cases, Deaths) %>%
mutate(ActiveCases=Cases-Deaths, FatalityRate=(Deaths/Cases)*1000) %>%
arrange(desc(Date))
South_Korea<- as_tibble(South_Korea)
South_Korea
South_Korea %>% ggplot(aes("", FatalityRate, color=as.factor(Month)))+
geom_point()+
geom_jitter()+
facet_grid(~Month, scales="free")+
labs(title="Güney Korede'ki Fatalite Hızlarının Aylara Göre Dağılımı")+
xlab("") + ylab("Fatalite Hızı (Binde)")+
labs(caption = "Analyzed by Tevfik Bulut, Data Source:https://www.ecdc.europa.eu")+
theme(plot.caption = element_text(color = "blue", face="italic", hjust=0.5)) +
theme(legend.position = "none")
South_Koreafrmonth<-select(South_Korea, Month, Cases, Deaths) %>%
group_by(Month) %>%
summarise(CasesSum = sum(Cases), DeathsSum = sum(Deaths), FatalityRate= round(sum(Deaths)/sum(Cases)*1000,1))
formattable(South_Koreafrmonth,
align = rep("l",4),
list('Date' = formatter("span", style = ~ style(color = "grey", font.weight = "bold")),
'FatalityRate' = color_bar("#FA614B")))
german<-df %>%
filter(Cases>0, Country=="Germany") %>%
group_by(Date, Month, Country, Cases, Deaths) %>%
mutate(ActiveCases=Cases-Deaths, FatalityRate=(Deaths/Cases)*1000) %>%
arrange(desc(Date))
german<- as_tibble(german)
german
german %>% ggplot(aes("", FatalityRate, color=as.factor(Month)))+
geom_point()+
geom_jitter()+
facet_grid(~Month, scales="free")+
labs(title="Almanya'daki Fatalite Hızlarının Aylara Göre Dağılımı")+
xlab("") + ylab("Fatalite Hızı (Binde)")+
labs(caption = "Analyzed by Tevfik Bulut, Data Source:https://www.ecdc.europa.eu")+
theme(plot.caption = element_text(color = "blue", face="italic", hjust=0.5)) +
theme(legend.position = "none")
germanfrmonth<-select(german, Month, Cases, Deaths) %>%
group_by(Month) %>%
summarise(CasesSum = sum(Cases), DeathsSum = sum(Deaths), FatalityRate= round(sum(Deaths)/sum(Cases)*1000,2))
formattable(germanfrmonth,
align = rep("l",4),
list('Date' = formatter("span", style = ~ style(color = "grey", font.weight = "bold")),
'FatalityRate' = color_bar("#FA614B")))
top<-df %>%
filter(Cases>0) %>%
group_by(Date, Month, Country, Cases, Deaths) %>%
mutate(ActiveCases=Cases-Deaths, FatalityRate=(Deaths/Cases)*1000) %>%
arrange(desc(Month))
top<- as_tibble(top)
top
topfrmonth<-select(top, Country, Cases, Deaths) %>%
group_by(Country) %>%
summarise(CasesSum = sum(Cases), DeathsSum = sum(Deaths), FatalityRate= round((sum(Deaths)/sum(Cases)*1000),1))%>%
arrange(desc(FatalityRate)) %>%
top_n(12)
formattable(topfrmonth,
align = rep("l",4),
list('Date' = formatter("span", style = ~ style(color = "grey", font.weight = "bold")),
'FatalityRate' = color_bar("#66CDAA")))
#####################################################
bottom<-df %>%
filter(Cases>0) %>%
group_by(Date, Month, Country, Cases, Deaths) %>%
mutate(ActiveCases=Cases-Deaths, FatalityRate=(Deaths/Cases)*1000) %>%
arrange(desc(Month))
bottom<- as_tibble(bottom)
bottom
bottomfrmonth<-select(bottom, Country, Cases, Deaths) %>%
group_by(Country) %>%
summarise(CasesSum = sum(Cases), DeathsSum = sum(Deaths), FatalityRate= round((sum(Deaths)/sum(Cases)*1000),1)) %>%
arrange(desc(FatalityRate)) %>%
top_n(-12)
formattable(bottomfrmonth,
align = rep("l",4"),
list('Date' = formatter("span", style = ~ style(color = "grey", font.weight = "bold")),
'FatalityRate' = color_bar("#FA8072")))
usa<-df %>%
filter(Cases>0, Country=="United_States_of_America") %>%
group_by(Date, Month, Country, Cases, Deaths) %>%
mutate(ActiveCases=Cases-Deaths, FatalityRate=(Deaths/Cases)*1000) %>%
arrange(desc(Date))
usa<- as_tibble(usa)
usa
usa %>% ggplot(aes("", FatalityRate, color=as.factor(Month)))+
geom_point()+
geom_jitter()+
facet_grid(~Month, scales="free")+
labs(title="Amerika'daki Fatalite Hızlarının Aylara Göre Dağılımı")+
xlab("") + ylab("Fatalite Hızı (Binde)")+
labs(caption = "Analyzed by Tevfik Bulut, Data Source:https://www.ecdc.europa.eu")+
theme(plot.caption = element_text(color = "blue", face="italic", hjust=0.5)) +
theme(legend.position = "none")
usafrmonth<-select(usa, Month, Cases, Deaths) %>%
group_by(Month) %>%
summarise(CasesSum = sum(Cases), DeathsSum = sum(Deaths), FatalityRate= round(sum(Deaths)/sum(Cases)*1000,1))
formattable(usafrmonth,
align = rep("l",4),
list('Date' = formatter("span", style = ~ style(color = "grey", font.weight = "bold")),
'FatalityRate' = color_bar("#FA614B")))
uk<-df %>%
filter(Cases>0, Country=="United_Kingdom") %>%
group_by(Date, Month, Country, Cases, Deaths) %>%
mutate(ActiveCases=Cases-Deaths, FatalityRate=(Deaths/Cases)*1000) %>%
arrange(desc(Date))
uk<- as_tibble(uk)
uk
uk %>% ggplot(aes("", FatalityRate, color=as.factor(Month)))+
geom_point()+
geom_jitter()+
facet_grid(~Month, scales="free")+
labs(title="İngiltere'deki Fatalite Hızlarının Aylara Göre Dağılımı")+
xlab("") + ylab("Fatalite Hızı (Binde)")+
labs(caption = "Analyzed by Tevfik Bulut, Data Source:https://www.ecdc.europa.eu")+
theme(plot.caption = element_text(color = "blue", face="italic", hjust=0.5)) +
theme(legend.position = "none")
ukfrmonth<-select(uk, Month, Cases, Deaths) %>%
group_by(Month) %>%
summarise(CasesSum = sum(Cases), DeathsSum = sum(Deaths), FatalityRate= round(sum(Deaths)/sum(Cases)*1000,1))
formattable(ukfrmonth,
align = rep("l",4),
list('Date' = formatter("span", style = ~ style(color = "grey", font.weight = "bold")),
'FatalityRate' = color_bar("#FA614B")))