Giriş / Introduction
Coronavirüsler (CoV), soğuk algınlığından Orta Doğu Solunum Sendromu (MERS-CoV) ve Şiddetli Akut Solunum Sendromu (SARS-CoV) gibi daha ciddi hastalıklara kadar pek çok hastalığa neden olan büyük bir virüs ailesidir. Bu yeni virüse, yüzey çıkıntıları taç görünümünde olduğu için coronavirus adı verilmiş olup, bu virüs ailesi tek zincirli, pozitif polariteli, zarflı RNA virüsleridir.
Koronavirüs hastalığı (COVID-19), 2019 yılında keşfedilen ve daha önce insanlarda tanımlanmamış yeni bir suştur. Suş, bir bakteri veya virüsün farklı alt türlerinin, aralarında genetik farklılıklar bulunan grupları olarak adlandırılır.
Bu Koronavirüsler zoonotiktir, yani hayvanlar ve insanlar arasında bulaşırlar. Ayrıntılı araştırmalar SARS-CoV’nin misk kedilerinden insanlara ve MERS-CoV’un tek hörgüçlü develerden insanlara bulaştığını göstermiştir. Bilinen birkaç koronavirüs henüz insanları enfekte etmeyen hayvanlarla taşınmaktadır.
Enfeksiyon belirtileri arasında solunum semptomları, ateş, öksürük, nefes darlığı ve solunum güçlüğü bulunur. Daha ciddi vakalarda, enfeksiyon pnömoniye, ciddi akut solunum sendromuna, böbrek yetmezliğine ve hatta ölüme neden olabilir. Solunum sistemi hastalıklarından biri olan pnömoni ya da halk arasında zatürre bakteriler başta olmak üzere mikroorganizmalara bağlı olarak ortaya çıkan akciğer dokusunun iltihaplanmasıdır. Grip gibi bu hastalık da bağışıklık (immun) sistemini baskılayan hastalıklardan biridir. Özellikle ileri yaşlardaki bireylerde hem bağışıklık sisteminin yaşla birlikte yavaşlaması hem de yaşa ve/veya diğer nedenlere bağlı olarak kronik ve diğer hastalıkların görülmesi bu yaşlardaki bireylerde ölüm oranlarını artırabilmektedir. Diğer bir deyişle, bu enfeksiyon komplikasyon yaratarak ve mevcut diğer hastalıkları tetikleyerek bu yaş gruplarında diğer yaş gruplarına göre daha öldürücü olabilir.
Enfeksiyonun yayılmasını önlemek için standart öneriler arasında düzenli el yıkama, öksürme ve hapşırma sırasında ağız ve burnun kapatılması, et ve yumurtaların iyice pişirilmesi yer alır. Öksürme ve hapşırma gibi solunum yolu rahatsızlığı belirtileri gösteren kişilerle yakın temastan kaçınmak bu hastalıktan korunmak için atılacak önemli koruyucu tedbirlerden biridir.
Hastalıktan nasıl korunulacağı hakkında bilgi verdikten sonra bu hastalığın teşhisinde kullanılan ve adı son zamanlarda sık sık duyulan PCR testi hakkında bilgi vermek faydalı olacaktır. PCR, ingilizce adıyla “Polymerase Chain Reaction” kelimelerinin bir araya gelmesinden oluşan ve moleküler biyoloji alanında kullanılan bir tekniktir.Türkçe ise ise polimeraz zincir reaksiyonu olarak adlandırılır. Bazen PCR moleküler fotokopi (molecular photocopying) olarak da adlandırılabilmektedir. DNA’nın küçük bölümlerini kopyalamak için kullanılan hızlı ve ucuz bir tekniktir. Moleküler ve genetik analizler için büyük miktarlarda DNA örneği gerekli olduğundan, izole edilmiş DNA parçaları üzerinde çalışmalar PCR amplifikasyonu olmadan neredeyse imkansızdır. PCR’ın kullanım alanları şöyle özetlenebilir:
- Amplifiye edildikten sonra PCR ile üretilen DNA, birçok farklı laboratuvar prosedüründe kullanılabilir. Örneğin, İnsan Genom Projesindeki (HGP) çoğu haritalama tekniği PCR’a dayanmaktadır.
- Parmak izi, bakteri veya virüslerin (özellikle AIDS) tespiti ve genetik bozuklukların teşhisi gibi bir dizi laboratuvar ve klinik teknikte de değerlidir.
PCR ( Polimeraz Zincir Reaksiyonu) tekniğinin çalışma şeması ise aşağıdaki şekilde verilmiştir.
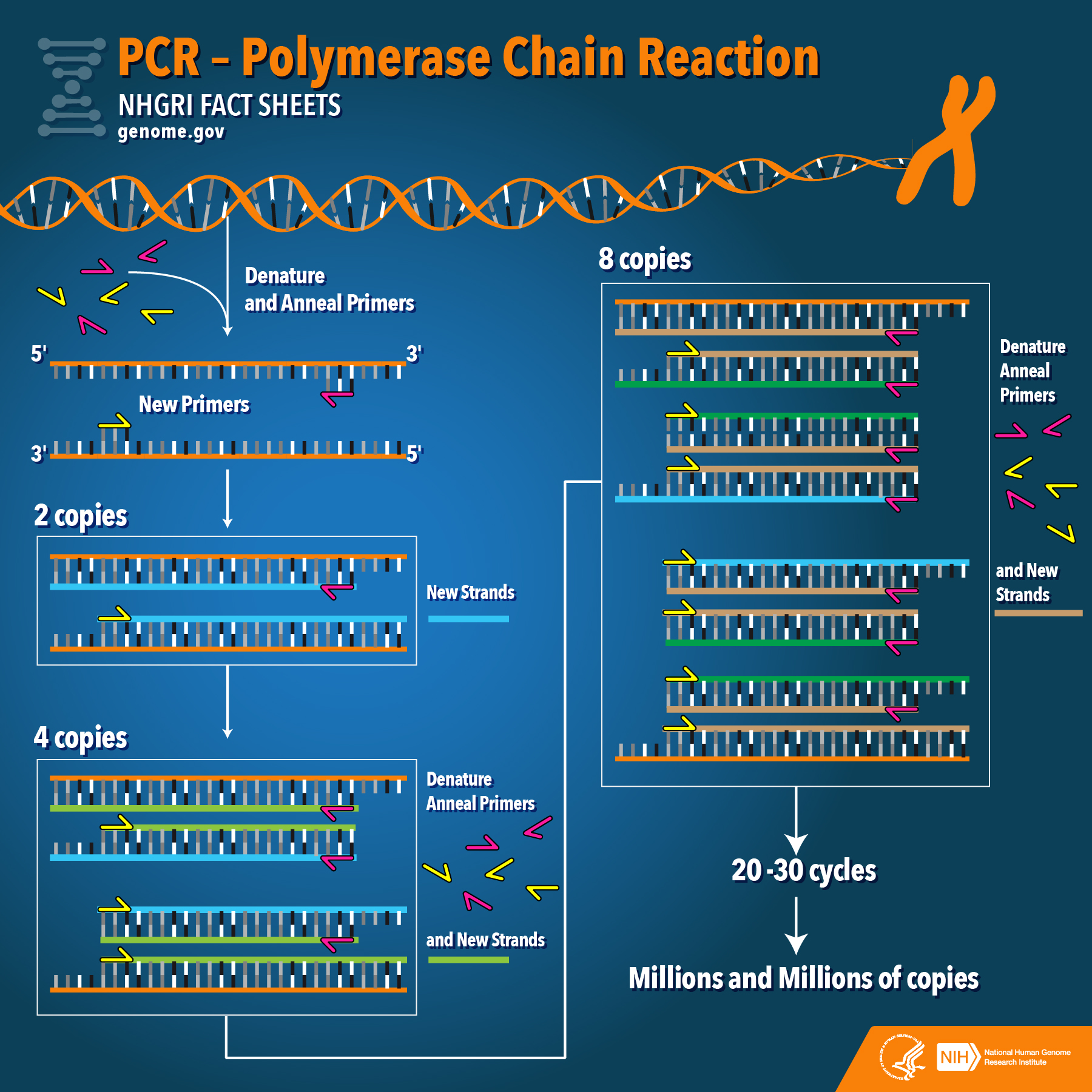
PCR tekniğini kullanarak koronavirüs teşhisinin nasıl konulduğuna ilişkin bir video aşağıda verilerek teşhis süreci hakkında bilgi verilmesi amaçlanmıştır.
AMBOSS: Medical Knowledge Distilled
Bilindiği üzere koronavirüs hastalığının teşhis ve özellikle tedavisine yönelik olarak ilaç ve aşı geliştirme alanında Dünya’da ve ülkemizde yoğun bir mesai harcanmaktadır. Bunlardan biri ülkemizde 02.04.2020 tarihinde Sanayi ve Teknoloji Bakanlığı ve TÜBİTAK bünyesinde “Covid-19 Türkiye Platformu Aşı ve İlaç Geliştirme” bilim insanların katıldığı sanal bir konferans gerçekleştirilmiştir. Türkiye dışında da benzer çalışmalar sanal ortamda çalıştay ve konferans olarak yapılmaktadır. Dünyada yapılan ve benim de katılım sağladığım önde giden bu çalışmalardan biri de Avrupa Öğrenme ve Akıllı Sistemler Laboratuvarı ingilizce adıyla European Laboratory for Learning and Intelligent Systems (ELLIS)’ın 02.04.2020 tarihinde düzenlediği “ELLIS Against Covid-19” adlı çalıştaydır. Dünyanın farklı üniversitelerinden bilim insanlarının katılım sağladığı çalıştayda koranavirüs pandemisinin teşhisinde kullanılan yapay zeka teknolojilerinden koronavirüsün tedavisinde kullanılan ilaçların geliştirilmesinde kullanılan yapay zeka teknolojilerine kadar pek çok konu ele alınmıştır. Çalıştayda ağırlıklı olarak ele alınan konu yapay zekanın teşhis ve tedavide nasıl kullanıldığı ve kullanılacağı idi. Çalıştayda koronavirüs ile mücadele çok güzel uygulama örneklerine yer verilmiştir. Daha önce de dile getirdiğim gibi yapay zeka günümüzün ve geleceğin en kritik teknolojisidir. Bu gerek 4. Sanayi Devrimi gerekse Toplum 5.0 açısından olsun böyledir ve farklı ülkeler yapay zekayı teşhis ve tedavi (robotik cerrahi ve ilaç geliştirme) alanlarında kullanmak için çok ciddi adımlar atmıştır. Bu yapıların da ülkemizde kurulması ve yapay zeka enstitülerinin ülkemizde yaygınlaştırılması gerekmektedir. Aynı zamanda Türkiye Büyük Millet Meclisi bünyesinde Yapay Zeka Komisyonu kurulmalıdır. Yapay zeka özünde öğrenen sistemler olduğu ve sürekli gelişim gösterdiği için sektör ayrımı olmaksızın her alanda kendine uygulama alanı bulan bir teknolojidir.
Koronavirüs salgını Çin ile birlikte diğer ülkelere de yayıldığı için Dünya Sağlık Örgütü tarafından 30/01/2020 tarihinde uluslararası halk sağlığı açısından acil durum ilan edilmiştir. Bu hastalıktan bahsedilirken lise ve üniversite yıllarımda benim de ders olarak aldığım Epidemiyoloji biliminden bahsetmemek olmaz. Epidemiyoloji, toplumda sağlıkla ilgili durumların veya olayların (hastalık dahil) dağılımını, görülme sıklıklarını ve bunları etkileyen faktörleri inceleyen bir tıp bilimi dalıdır.
Epidemiyolojik araştırmalar yapmak için çeşitli yöntemler kullanılabilir. Bu yöntemler dağılımı incelemek için sürveyans, tanımlayıcı çalışmalar ve hastalık belirleyicilerini incelemek için analitik çalışmalar olabilir. Bilindiği üzere Koronavirüs hastalığı artık pandemik bir vaka olarak adlandırılmaktadır. Epidemiyolojinin konu alanına giren pandemi dünyada birden fazla ülkede veya kıtada, çok geniş bir alanda
yayılan ve etkisini gösteren salgın hastalıklara verilen genel isim olarak ifade edilmektedir. Diğer bir deyişle, salgın (epidemi) durumundaki bir hastalığın ülke sınırlarını aşarak diğer ülkelere yayılım göstermesidir.
Avrupa Birliği kurumlarından biri olan “Avrupa Hastalık Önleme ve Kontrol Merkezi (European Centre for Disease Prevention and Control)”‘ne göre ön bulgular koronavirüs hastalığından vakalara dayalı ölüm oranı (fatalite hızı)’nın binde 20-30 olduğunu göstermektedir. Bu, 2003 SARS salgınından önemli ölçüde daha az. Bununla birlikte, mevsimsel gripin neden ölüm oranlarından çok daha yüksektir.
Çalışma kapsamında koronavirüs pandemi vakalarının epidemiyolojik hızlarla birlikte keşifsel veri analizi (EDA) yapılarak vakaların izlediği seyrin ortaya konulması amaçlanmıştır
Metodoloji / Methodology
Çalışmanın amacı, koronavirüs vakalarının epidemiyolojik düzlemde Türkiye’de ve diğer ülkelerde izlediği seyri detaylı bir şekilde keşifsel veri analiziyle ortaya konulara karar vericilere katkı sunmaktır.
Koronavirüs vakalarının analizinde Türkiye veri setinin oluşturulmasında Türkiye İstatistik Kurumu (TÜİK)’ndan ve Dünya Sağlık Örgütü (DSÖ) raporlarından ve Sağlık Bakanlığı basın açıklamalarından yararlanılmıştır. Bunun nedeni Türkiye veri setine daha derinlemesine bakılmak istenmesidir.
Çalışma kapsamında diğer ülkelere ilişkin koronavirüs vakaları ise Johns Hopkins Üniversitesi Sistem Bilimi ve Mühendisliği Merkezi (JHU CCSE) tarafından desteklenen “The Humanitarian Data Exchange (HDX) (İnsani Veri Alışverişi)” adlı platforma ait https://data.humdata.org web sitesinden alınmıştır. Önceki çalışmalardan faklı olarak bu platformdan veri alınmasının nedeni veri setleri içerisinde onaylanmış ve ölüm vakaları dışında iyileşen koronavirüs vakalarına da yer verilmiş olmasıdır.
Analizlerde Türkiye verisi için Microsoft Excel 2016 ve diğer ülke verileri için R programlama dili kullanılmıştır.
Keşifsel veri analizine geçilmeden önce vaka sayılarından yararlanarak epidemiyolojik hızlar hesaplanmıştır. Bu nedenle, çalışmada kullanılacak hızları açıklamanın faydalı olacağı düşünülmektedir.
Epidemiyoloji biliminde hız kavramı, bir sağlık olayının nüfus içindeki görülme frekansını hesaplamada kullanılan bir ölçüttür. Hızlar, belirli bir zaman diliminde meydana gelen olayların sayısının olayın meydana geldiği nüfusa bölünerek hesaplanır. Eşitliğin payı, sağlık olayı ile karşılaşanların sayısını (X), payda ise risk altındaki nüfusu (X+Y) göstermektedir. Risk altındaki nüfus hastalığa veya sağlık olaylarına karşı duyarlı olan nüfusu ifade etmektedir. Formülize edilirse;
Hız (Rate) = (X / X +Y) * k
Burada, k katsayıyı göstermektedir.
Analizde hızlardan girdi değişkeni olarak kullanılması düşünülen ilk gösterge ölüm veya mortalite ölçütlerinden biri olan Kaba Ölüm Hızı‘dır. Bu hız nüfusun sağlık düzeyini ortaya koymada duyarlı olmasa da bir fikir vermesi açısından önemlidir. Kaba ölüm hızı aşağıdaki eşitlikte gösterilmiştir.
Kaba Ölüm Hızı (Crude Death Rate) = (Bir toplumda belirli bir süre içinde meydana gelen ölüm sayısı / Aynı toplumun aynı süredeki
(yıl ortası) nüfusu) * 1000
Crude death rate = (Total number of deaths during a given time interval / Mid-interval population) * 1000
Girdi değişkeni olarak kullanılması düşünülen ikinci gösterge ölüm veya fatalite ölçütlerinden biri olan Fatalize Hızı‘dır. Bu gösterge kaba ölüm hızına göre daha duyarlı bir göstergedir. Diğer bir ifadeyle, hastalığa yakalananların ne kadarının öldüğünü gösterir. Fatalite hızı aşağıdaki eşitlik yardımıyla hesaplanmaktadır;
Fatalite Hızı (Fatality Rate) = (Nüfusta belirli bir süre içinde “X” hastalığından ölenlerin sayısı / Aynı nüfusun aynı süre içinde “X” hastalığına yakalananların sayısı) * 100
Fatality Rate = (Number of cause-specific deaths among the incident cases / Total number of incident cases) * 100
Türkiye ve Dünya Örnekleri / Cases of Turkey and World Countries
Analizde kullanılan veri Birleşmiş Milletler İnsani İşlerin Koordinasyon Bürosu (OCHA)‘nun web sitelerinden biri olan İnsani Veri Alışverişi (HDX) web sitesinden csv (Comma-seperated value) uzantılı olarak okutulan onaylanmış, ölen ve iyileşen koronavirüs vaka sayılarını içeren 3 farklı veri setinden oluşmaktadır.
Çalışmanın bu kısmında epidemiyolojik hızlarla birlikte keşifsel veri analizi yapılarak Dünyada koronavirüs vakalarıyla ilgili olarak öne çıkan ülkeler verilmiştir.
Aşağıda yazdığım Python kod bloğu ile birlikte “İnsani Veri Alışverişi (HDX)” veri tabanından güncel veri akışı sağlanabilecektir. Grafiklerde ise R programlama dili kullanılmıştır. Aşağıdaki kod bloğunda görüleceği üzere mevcut veri setlerinde olmayan 1 adet değişken hesaplanmıştır. Daha sonra bu değişkenler kod bloğu ile veri akışı sağlanmasından sonra birleştirilerek oluşturulan yeni veri setine ilave edilmiştir. İlave edilen değişkenler şöyledir:
- Fatali Fızı (Case Fatality Rate:CFR): Onaylanmış vaka sayılarının ölüm vaka sayılarına bölünmesiyle hesaplanmıştır. Çarpım katsayısı (k) ise 1000 olarak alınmıştır.
import numpy as np
import scipy as sp
import pandas as pd
import matplotlib as mpl
import seaborn as sns
case = pd.read_csv("https://data.humdata.org/hxlproxy/data/download/time_series_covid19_confirmed_global_narrow.csv?dest=data_edit&filter01=merge&merge-url01=https%3A%2F%2Fdocs.google.com%2Fspreadsheets%2Fd%2Fe%2F2PACX-1vTglKQRXpkKSErDiWG6ycqEth32MY0reMuVGhaslImLjfuLU0EUgyyu2e-3vKDArjqGX7dXEBV8FJ4f%2Fpub%3Fgid%3D1326629740%26single%3Dtrue%26output%3Dcsv&merge-keys01=%23country%2Bname&merge-tags01=%23country%2Bcode%2C%23region%2Bmain%2Bcode%2C%23region%2Bsub%2Bcode%2C%23region%2Bintermediate%2Bcode&filter02=merge&merge-url02=https%3A%2F%2Fdocs.google.com%2Fspreadsheets%2Fd%2Fe%2F2PACX-1vTglKQRXpkKSErDiWG6ycqEth32MY0reMuVGhaslImLjfuLU0EUgyyu2e-3vKDArjqGX7dXEBV8FJ4f%2Fpub%3Fgid%3D398158223%26single%3Dtrue%26output%3Dcsv&merge-keys02=%23adm1%2Bname&merge-tags02=%23country%2Bcode%2C%23region%2Bmain%2Bcode%2C%23region%2Bsub%2Bcode%2C%23region%2Bintermediate%2Bcode&merge-replace02=on&merge-overwrite02=on&filter03=explode&explode-header-att03=date&explode-value-att03=value&filter04=rename&rename-oldtag04=%23affected%2Bdate&rename-newtag04=%23date&rename-header04=Date&filter05=rename&rename-oldtag05=%23affected%2Bvalue&rename-newtag05=%23affected%2Binfected%2Bvalue%2Bnum&rename-header05=Value&filter06=clean&clean-date-tags06=%23date&filter07=sort&sort-tags07=%23date&sort-reverse07=on&filter08=sort&sort-tags08=%23country%2Bname%2C%23adm1%2Bname&tagger-match-all=on&tagger-default-tag=%23affected%2Blabel&tagger-01-header=province%2Fstate&tagger-01-tag=%23adm1%2Bname&tagger-02-header=country%2Fregion&tagger-02-tag=%23country%2Bname&tagger-03-header=lat&tagger-03-tag=%23geo%2Blat&tagger-04-header=long&tagger-04-tag=%23geo%2Blon&header-row=1&url=https%3A%2F%2Fraw.githubusercontent.com%2FCSSEGISandData%2FCOVID-19%2Fmaster%2Fcsse_covid_19_data%2Fcsse_covid_19_time_series%2Ftime_series_covid19_confirmed_global.csv")
case = case.drop(case.index[[0]])
case = case.iloc[:,[1,2,3,4,5]]
case = case.rename(columns={"Country/Region": "Ulke","Date":"Tarih","Value": "Vaka", "Lat":"Lat","Long":"Long"})
case = case.astype({'Vaka': 'int64','Tarih': 'datetime64[ns]', 'Lat': 'float64', 'Long':'float64'})
case
url1="https://data.humdata.org/hxlproxy/data/download/time_series_covid19_deaths_global_narrow.csv?dest=data_edit&filter01=merge&merge-url01=https%3A%2F%2Fdocs.google.com%2Fspreadsheets%2Fd%2Fe%2F2PACX-1vTglKQRXpkKSErDiWG6ycqEth32MY0reMuVGhaslImLjfuLU0EUgyyu2e-3vKDArjqGX7dXEBV8FJ4f%2Fpub%3Fgid%3D1326629740%26single%3Dtrue%26output%3Dcsv&merge-keys01=%23country%2Bname&merge-tags01=%23country%2Bcode%2C%23region%2Bmain%2Bcode%2C%23region%2Bsub%2Bcode%2C%23region%2Bintermediate%2Bcode&filter02=merge&merge-url02=https%3A%2F%2Fdocs.google.com%2Fspreadsheets%2Fd%2Fe%2F2PACX-1vTglKQRXpkKSErDiWG6ycqEth32MY0reMuVGhaslImLjfuLU0EUgyyu2e-3vKDArjqGX7dXEBV8FJ4f%2Fpub%3Fgid%3D398158223%26single%3Dtrue%26output%3Dcsv&merge-keys02=%23adm1%2Bname&merge-tags02=%23country%2Bcode%2C%23region%2Bmain%2Bcode%2C%23region%2Bsub%2Bcode%2C%23region%2Bintermediate%2Bcode&merge-replace02=on&merge-overwrite02=on&filter03=explode&explode-header-att03=date&explode-value-att03=value&filter04=rename&rename-oldtag04=%23affected%2Bdate&rename-newtag04=%23date&rename-header04=Date&filter05=rename&rename-oldtag05=%23affected%2Bvalue&rename-newtag05=%23affected%2Binfected%2Bvalue%2Bnum&rename-header05=Value&filter06=clean&clean-date-tags06=%23date&filter07=sort&sort-tags07=%23date&sort-reverse07=on&filter08=sort&sort-tags08=%23country%2Bname%2C%23adm1%2Bname&tagger-match-all=on&tagger-default-tag=%23affected%2Blabel&tagger-01-header=province%2Fstate&tagger-01-tag=%23adm1%2Bname&tagger-02-header=country%2Fregion&tagger-02-tag=%23country%2Bname&tagger-03-header=lat&tagger-03-tag=%23geo%2Blat&tagger-04-header=long&tagger-04-tag=%23geo%2Blon&header-row=1&url=https%3A%2F%2Fraw.githubusercontent.com%2FCSSEGISandData%2FCOVID-19%2Fmaster%2Fcsse_covid_19_data%2Fcsse_covid_19_time_series%2Ftime_series_covid19_deaths_global.csv"
death= pd.read_csv(url1)
death = death.drop(death.index[[0]])
death = death.iloc[:,[1,2,3,4,5]]
death = death.rename(columns={"Country/Region": "Ulke","Date":"Tarih","Value": "Olen", "Lat":"Lat","Long":"Long"})
death = death.astype({'Olen': 'int32', 'Tarih': 'datetime64[ns]', 'Lat': 'float64', 'Long':'float64'})
death
url2="https://data.humdata.org/hxlproxy/data/download/time_series_covid19_recovered_global_narrow.csv?dest=data_edit&filter01=merge&merge-url01=https%3A%2F%2Fdocs.google.com%2Fspreadsheets%2Fd%2Fe%2F2PACX-1vTglKQRXpkKSErDiWG6ycqEth32MY0reMuVGhaslImLjfuLU0EUgyyu2e-3vKDArjqGX7dXEBV8FJ4f%2Fpub%3Fgid%3D1326629740%26single%3Dtrue%26output%3Dcsv&merge-keys01=%23country%2Bname&merge-tags01=%23country%2Bcode%2C%23region%2Bmain%2Bcode%2C%23region%2Bsub%2Bcode%2C%23region%2Bintermediate%2Bcode&filter02=merge&merge-url02=https%3A%2F%2Fdocs.google.com%2Fspreadsheets%2Fd%2Fe%2F2PACX-1vTglKQRXpkKSErDiWG6ycqEth32MY0reMuVGhaslImLjfuLU0EUgyyu2e-3vKDArjqGX7dXEBV8FJ4f%2Fpub%3Fgid%3D398158223%26single%3Dtrue%26output%3Dcsv&merge-keys02=%23adm1%2Bname&merge-tags02=%23country%2Bcode%2C%23region%2Bmain%2Bcode%2C%23region%2Bsub%2Bcode%2C%23region%2Bintermediate%2Bcode&merge-replace02=on&merge-overwrite02=on&filter03=explode&explode-header-att03=date&explode-value-att03=value&filter04=rename&rename-oldtag04=%23affected%2Bdate&rename-newtag04=%23date&rename-header04=Date&filter05=rename&rename-oldtag05=%23affected%2Bvalue&rename-newtag05=%23affected%2Binfected%2Bvalue%2Bnum&rename-header05=Value&filter06=clean&clean-date-tags06=%23date&filter07=sort&sort-tags07=%23date&sort-reverse07=on&filter08=sort&sort-tags08=%23country%2Bname%2C%23adm1%2Bname&tagger-match-all=on&tagger-default-tag=%23affected%2Blabel&tagger-01-header=province%2Fstate&tagger-01-tag=%23adm1%2Bname&tagger-02-header=country%2Fregion&tagger-02-tag=%23country%2Bname&tagger-03-header=lat&tagger-03-tag=%23geo%2Blat&tagger-04-header=long&tagger-04-tag=%23geo%2Blon&header-row=1&url=https%3A%2F%2Fraw.githubusercontent.com%2FCSSEGISandData%2FCOVID-19%2Fmaster%2Fcsse_covid_19_data%2Fcsse_covid_19_time_series%2Ftime_series_covid19_recovered_global.csv"
r = pd.read_csv(url2)
r = r.drop(r.index[[0]])
r = r.iloc[:,[1,2,3,4,5]]
r = r.rename(columns={"Country/Region": "Ulke","Date":"Tarih","Value": "Iyilesen", "Lat":"Lat","Long":"Long"})
r = r.astype({'Iyilesen': 'int32', 'Tarih': 'datetime64[ns]', 'Lat': 'float64', 'Long':'float64'})
r
#Veri Setlerinin Birleştirilmesi
x = case.merge(death, on=['Ulke','Lat','Long', 'Tarih'], how='left')
y = x.merge(r, on=['Ulke','Lat','Long', 'Tarih'], how='left')
result =result =y[(y.Vaka >0)]
result
result.isna().sum()# Variable: Iyilesen including 487 missing data
result=result.fillna(0)#it is assigned to 0 in place of NAN values
result
result.isna().sum()#Iyilesen: no missing data
result.info()#str gibi R'da olan: It give us idea about structure of data set
Veri setindeki değişken sayısı 7, gözlem sayısı ise 20876’dir. Aşağıda mevcut değişkenler, değişkenlerin veri tipi ile birlikte gözlem sayısı görülmektedir. Daha sonra yedi olan değişken sayısı fatalite hızı ilave edilerek 8’e çıkarılmıştır. Ancak mükerrer tarihler benzersiz tarihlere düşürüldükten kalan gözlem sayısı 10734’tür.
<class 'pandas.core.frame.DataFrame'>
Int64Index: 10734 entries, 0 to 20876
Data columns (total 8 columns):
Ulke 10734 non-null object
Lat 10734 non-null float64
Long 10734 non-null float64
Tarih 10734 non-null datetime64[ns]
Vaka 10734 non-null int64
Olen 10734 non-null float64
Iyilesen 10734 non-null float64
CFR 10734 non-null float64
dtypes: datetime64[ns](1), float64(5), int64(1), object(1)
memory usage: 754.7+ KB
Veri setinde koronavirüs salgınının yaşandığı aşağıdaki listesi verilen 184 ülke ve/veya bölge bulunmaktadır.
['Afghanistan',
'Albania',
'Algeria',
'Andorra',
'Angola',
'Antigua and Barbuda',
'Argentina',
'Armenia',
'Australia',
'Austria',
'Azerbaijan',
'Bahamas',
'Bahrain',
'Bangladesh',
'Barbados',
'Belarus',
'Belgium',
'Belize',
'Benin',
'Bhutan',
'Bolivia',
'Bosnia and Herzegovina',
'Botswana',
'Brazil',
'Brunei',
'Bulgaria',
'Burkina Faso',
'Burma',
'Burundi',
'Cabo Verde',
'Cambodia',
'Cameroon',
'Canada',
'Central African Republic',
'Chad',
'Chile',
'China',
'Colombia',
'Congo (Brazzaville)',
'Congo (Kinshasa)',
'Costa Rica',
"Cote d'Ivoire",
'Croatia',
'Cuba',
'Cyprus',
'Czechia',
'Denmark',
'Diamond Princess',
'Djibouti',
'Dominica',
'Dominican Republic',
'Ecuador',
'Egypt',
'El Salvador',
'Equatorial Guinea',
'Eritrea',
'Estonia',
'Eswatini',
'Ethiopia',
'Fiji',
'Finland',
'France',
'Gabon',
'Gambia',
'Georgia',
'Germany',
'Ghana',
'Greece',
'Grenada',
'Guatemala',
'Guinea',
'Guinea-Bissau',
'Guyana',
'Haiti',
'Holy See',
'Honduras',
'Hungary',
'Iceland',
'India',
'Indonesia',
'Iran',
'Iraq',
'Ireland',
'Israel',
'Italy',
'Jamaica',
'Japan',
'Jordan',
'Kazakhstan',
'Kenya',
'Korea, South',
'Kosovo',
'Kuwait',
'Kyrgyzstan',
'Laos',
'Latvia',
'Lebanon',
'Liberia',
'Libya',
'Liechtenstein',
'Lithuania',
'Luxembourg',
'MS Zaandam',
'Madagascar',
'Malawi',
'Malaysia',
'Maldives',
'Mali',
'Malta',
'Mauritania',
'Mauritius',
'Mexico',
'Moldova',
'Monaco',
'Mongolia',
'Montenegro',
'Morocco',
'Mozambique',
'Namibia',
'Nepal',
'Netherlands',
'New Zealand',
'Nicaragua',
'Niger',
'Nigeria',
'North Macedonia',
'Norway',
'Oman',
'Pakistan',
'Panama',
'Papua New Guinea',
'Paraguay',
'Peru',
'Philippines',
'Poland',
'Portugal',
'Qatar',
'Romania',
'Russia',
'Rwanda',
'Saint Kitts and Nevis',
'Saint Lucia',
'Saint Vincent and the Grenadines',
'San Marino',
'Sao Tome and Principe',
'Saudi Arabia',
'Senegal',
'Serbia',
'Seychelles',
'Sierra Leone',
'Singapore',
'Slovakia',
'Slovenia',
'Somalia',
'South Africa',
'South Sudan',
'Spain',
'Sri Lanka',
'Sudan',
'Suriname',
'Sweden',
'Switzerland',
'Syria',
'Taiwan*',
'Tanzania',
'Thailand',
'Timor-Leste',
'Togo',
'Trinidad and Tobago',
'Tunisia',
'Turkey',
'US',
'Uganda',
'Ukraine',
'United Arab Emirates',
'United Kingdom',
'Uruguay',
'Uzbekistan',
'Venezuela',
'Vietnam',
'West Bank and Gaza',
'Western Sahara',
'Zambia',
'Zimbabwe']
Dünya
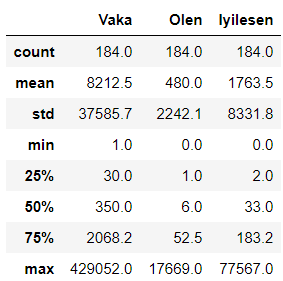
Dünyada görülen koronavirüs vakalarının tanımlayıcı istatistikleri aşağıdaki tabloda verilmiştir. Veri setindeki vaka sayıları kümülatif olarak ilerlemektedir.
Dünyadaki Koronavirüs Vakalarının Tanımlayıcı İstatistikleri
Kümülatif vaka sayılarına göre en yüksek 10 ülke aşağıdaki tabloda karşılaştırmalı olarak verilmiştir.
En Yüksek Kümülatif Vaka Sayısına Sahip 10 Ülke

En Yüksek Kümülatif Ölüm Vaka Sayısına Sahip 10 Ülke

En Yüksek Kümülatif İyileşen Vaka Sayısına Sahip 10 Ülke

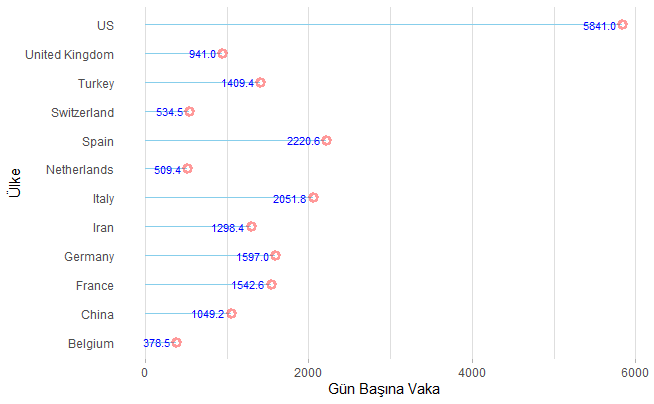
Vaka Sayısı En Yüksek 10 Ülkenin Gün Başına Vaka Sayıları
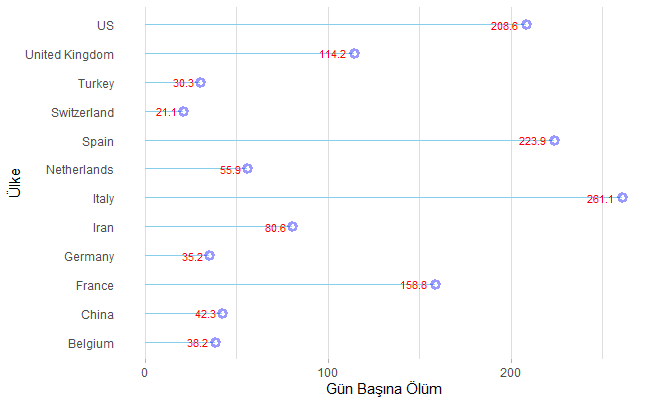
Vaka Sayısı En Yüksek 10 Ülkenin Gün Başına Ölüm Sayıları
Vaka Sayısı En Yüksek 10 Ülkenin Gün Başına İyileşen Sayıları

Dünya Ülkelerinin Gün Başına Vaka, Ölüm ve İyileşen İstatistikleri
Aşağıdaki linkten 184 ülkenin gün başına vaka, ölüm ve iyileşen istatistiklerini indirebilirsiniz. Burada hangi ülkenin kaç günde ne kadar vaka, ölüm ve iyileşen sayısına ulaştığı görülebilir.
Sonuç / Conclusion
Özetle, İnsani Veri Alışverişi (HDX) platformundan alınan 3 farklı koronavirüs hastalık veri setinin birleştirilmesinden sonra epidemiyolojik hızlarla birlikte keşifsel veri analizi yapılarak koronavirüs pandemisinin trendi hakkında farkındalık oluşturulması amaçlanmıştır.
Faydalı olması dileğiyle.
Sağlıkla ve bilimle kalınız.
Saygılarımla.
Note: It can not be cited or copied without referencing.
Not: Kaynak gösterilmeden alıntı yapılamaz veya kopyalanamaz.
Yararlanılan Kaynaklar / Resources Utilized
- World Health Organization (2020). Report of the WHO-China Joint Mission on Coronavirus Disease 2019 (COVID-19). Available online at: https://www.who.int/docs/default-source/coronaviruse/who-china-joint-mission-on-covid-19-final-report.pdf
- https://www.python.org/
- https://www.who.int/health-topics/coronavirus
- https://www.ecdc.europa.eu/en/publications-data/download-todays-data-geographic-distribution-covid-19-cases-worldwide
- http://www.hastane.hacettepe.edu.tr/_948.html
- https://www.youtube.com/watch?v=mOV1aBVYKGA&t=42s
- Humanitarian Data Exchange (HDX), https://data.humdata.org/. 19.03.2020
- http://www.halksagligi.hacettepe.edu.tr/sunumlar_ve_seminerler/OLCUTLERTB18MART2015.pdf
- https://www.youtube.com/watch?v=4KFP3tay5vM
- De Livera, A. M., Hyndman, R. J., & Snyder, R. D. (2011). Forecasting time series with complex seasonal patterns using exponential smoothing. J American Statistical Association, 106(496), 1513–1527. https://robjhyndman.com/publications/complex-seasonality/
- http://tagteam.harvard.edu/hub_feeds/1981/feed_items/273923
- https://robjhyndman.com/papers/ComplexSeasonality.pdf
- https://www.medicalpark.com.tr/zaturre-nedir-belirtileri-ve-tedavileri-nelerdir/hg-152
- https://www.who.int/topics/epidemiology/en/
- https://www.grip.gov.tr/depo/saglik-calisanlari/ulusal_pandemi_plani.pdf
- https://www.who.int/docs/default-source/coronaviruse/getting-workplace-ready-for-covid-19.pdf
- https://www.cdc.gov/csels/dsepd/ss1978/lesson3/section3.html
- https://www.genome.gov/about-genomics/fact-sheets/Polymerase-Chain-Reaction-Fact-Sheet
- https://www.yourgenome.org/facts/what-is-pcr-polymerase-chain-reaction
- https://go.amboss.com/covid-swab
- https://www.youtube.com/watch?v=syXd7kgLSN8
- https://ellis.eu/